gène


Segment d'ADN conditionnant la synthèse d'une ou de plusieurs protéines et, donc, la manifestation et la transmission d'un caractère héréditaire déterminé.
On trouve des gènes chez tous les êtres vivants. Ce sont des segments de molécules d'ADN, sauf chez certains virus dont le matériel génétique est constitué d’ARN. Chez les procaryotes, ils peuvent être groupés en unités fonctionnelles appelés opérons (les gènes d’un opéron concourent à la réalisation d’une même fonction ; ils sont adjacents et transcrits en ARNm – ou inhibés – ensemble).
Chez les eucaryotes, les gènes sont essentiellement portés par les chromosomes contenus dans le noyau cellulaire, mais il en existe aussi dans les mitochondries et, chez les végétaux, dans les chloroplastes (ces gènes sont portés par les ADN respectivement mitochondrial et chloroplastique).
L’être humain possède environ 25 000 gènes différents, répartis sur 23 paires de chromosomes.
1. Le génotype détermine le phénotype
Les gènes sont situés à des endroits bien spécifiques des chromosomes. La localisation d’un gène sur un chromosome est appelée locus (au pluriel locus ou loci). Cette localisation est toujours identique d'une génération à la suivante. Les chromosomes allant par paires, chaque cellule possède chaque gène en double. Seuls les gènes portés par les chromosomes X et Y chez l'individu de sexe masculin sont uniques. L'ensemble des gènes d’un organisme ou d’un individu constitue son génotype (ne pas confondre avec le génome , qui est l'ensemble du matériel génétique, c'est-à-dire toutes les molécules d'ADN d'une cellule).
Les gènes gouvernent la synthèse d'ARN de plusieurs types, et à partir de là de protéines, qui sont directement ou indirectement responsables de tous les caractères, visibles ou invisibles, de l'organisme (le phénotype). Les gènes subissent spontanément des transformations, les mutations, qui peuvent aussi être induites par des rayonnements (rayons X, rayons ultraviolets) ou des substances chimiques et entraînent la modification du caractère déterminé par le gène .
→ génétique.
2. Les formes alléliques d'un gène
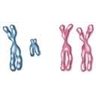
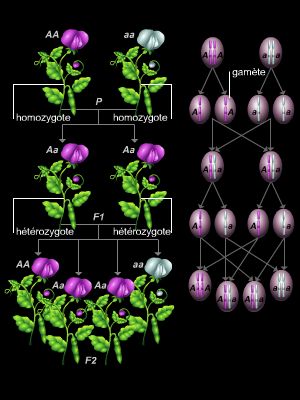
Les différentes versions d'un gène (le gène « couleur de la graine » chez le pois, par exemple) sont appelées allèles (graine verte, graine jaune). Lorsque l'allèle est le même sur les deux chromosomes, le sujet est dit homozygote. Si les deux allèles sont différents, il est dit hétérozygote. Si chaque allèle a subi une mutation différente, le sujet est dit double hétérozygote, ou hétérozygote composite. Certains caractères, comme la couleur des yeux, sont déterminés non par un seul gène, mais par plusieurs. Les effets des différents allèles des différents gènes se mélangent alors. C’est pourquoi des personnes aux yeux marrons peuvent par exemple posséder des allèles « bleus » masqués et avoir des enfants aux yeux bleus.
3. Dominance et récessivité
Chez un individu hétérozygote, seul se manifeste au niveau du phénotype le gène dit dominant. L’autre, appelé récessif, ne pourra s’exprimer que chez un individu homozygote pour cet allèle. Ainsi, si une maladie héréditaire qui se manifeste seulement si les deux allèles du gène en cause sont mutés, elle est dite à transmission récessive. Si, au contraire, un seul allèle muté suffit pour que la maladie se manifeste, elle est dite à transmission dominante.
Quand deux allèles (ou plus) s’expriment en même temps, on dit qu’ils sont codominants (c’est le cas par exemple des gènes qui codent pour la couleur de la peau).
Dans certains cas, des facteurs environnementaux peuvent influer sur l’expression des gènes et le résultat final au niveau du phénotype (c’est le cas par exemple de l’alimentation et de l’activité physique sur la taille ; → croissance).

Par convention, les allèles dominants sont notés par une lettre majuscule et les allèles récessifs par une minuscule. Un homozygote portant l’allèle dominant du gène A s’écrira (AA), un hétérozygote (Aa) et un homozygote pour l’allèle récessif (Aa). (Nota Bene : les génotypes se notent entre parenthèses, les phénotypes entre crochets).
4. Les gènes liés au sexe
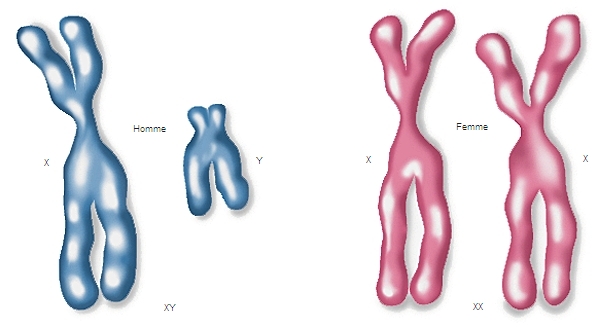
Chez les animaux, tous les chromosomes vont par paires d’homologues à l’exception d’une, la paire de chromosomes sexuels ou hétérosomes (chez les mammifères par exemple, les femelles sont XX, les mâles XY ; chez les oiseaux, les femelles sont ZW et les mâles ZZ). Ces chromosomes sont non seulement dissemblables, mais ils ne portent pas les mêmes gènes. Notamment, si chez les mammifères, le chromosome Y (dont la longueur correspond à environ un tiers du chromosome X) porte très peu de gènes. En fait, la plupart des gènes du chromosome X n’ont pas d’équivalent sur le chromosome Y. Ces gènes, dits gènes liés au sexe, se transmettent donc à la descendance selon un mode particulier : en effet les allèles récessifs s’expriment de façon directe, en un seul exemplaire (puisqu’ils n’ont pas d’autre allèle en face).
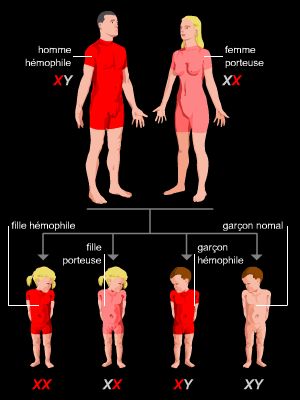
Beaucoup de maladies sont causées par des gènes liés au sexe, comme l’hémophilie et le daltonisme pour ne citer que les plus connues.
Pour en savoir plus, voir l'article sexe.
5. La transcription des gènes
5.1. Le déroulement de la transcription
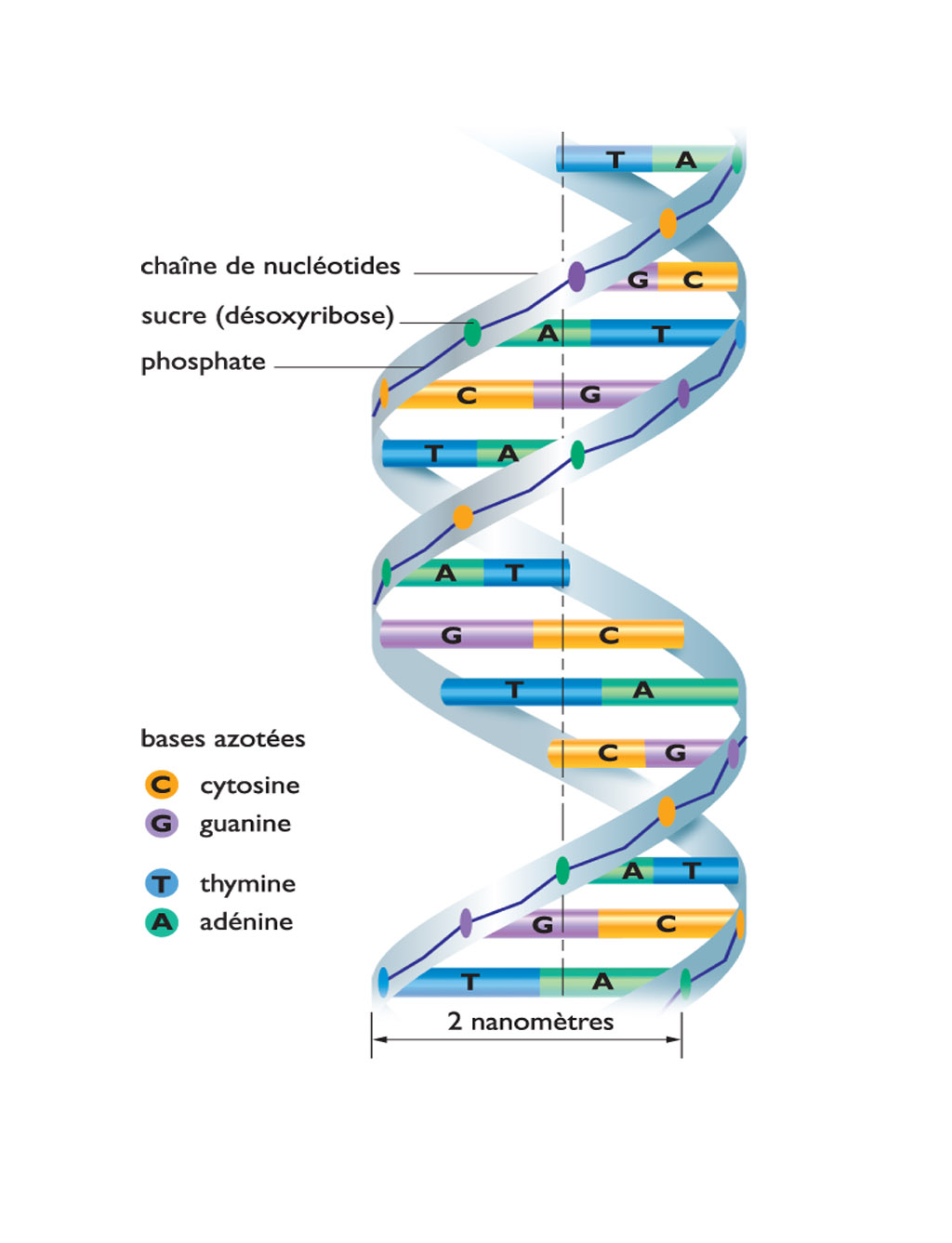
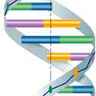
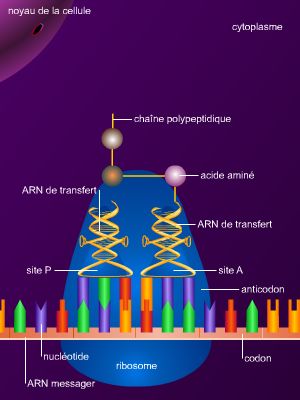
La transcription correspond à la synthèse d’une molécule d’ARN à partir d’un modèle ADN. L'ADN est constitué d'une suite linéaire de millions de nucléotides formés chacun de l'enchaînement d'un groupement phosphate et d'un sucre, le désoxyribose, portant une base pouvant être une adénine, une cytosine, une guanine ou une thymine (A, C, G ou T) ; l'information réside dans la suite des bases (la séquence nucléotidique). L'ADN comporte en réalité deux brins complémentaires de directions opposées enroulés l'un autour de l'autre (structure en double hélice élucidée par Watson et Crick en 1953). On peut la décrire comme un escalier en colimaçon, où les deux chaînes sucres-phosphates constituent les rampes, et les bases, tournées vers l'intérieur et se faisant face deux à deux, les marches. Dans l'ADN, une adénine s'apparie toujours avec une thymine, et une guanine toujours avec une cytosine (ces bases sont dites complémentaires entre elles) : on ne trouve donc que des « marches » A-T, T-A, C-G ou G-C. Elle constitue le fondement, non seulement de la perpétuation de l'information génétique (lors de la réplication), mais aussi de la transcription. Pendant celle-ci, la double hélice se déroule localement (au niveau du gène à transcrire, appelé région codante) et de façon transitoire. Une enzyme, l'ARN polymérase, se fixe en amont du gène (sur un site appelé promoteur), non sur le brin codant mais sur le brin opposé, qui va servir de matrice ; puis elle glisse le long du brin matrice en synthétisant un brin d'ARN messager complémentaire selon les règles de l’appariement : adénine-uracile (dans l’ARN, l’uracile remplace la thymine) et guanine-cytosine. Ainsi, l'ARN messager possède-t-il la même séquence que le brin codant d'ADN de départ, mais comporte des riboses au lieu des désoxyriboses et l'uracile (U) à la place de la thymine.

Chez les bactéries, les séquences promotrices qui déterminent le début de la transcription contiennent très souvent la séquence TTGACA, située à la distance d'environ 35 nucléotides en amont du point de départ du gène, et la séquence TATAAT, située à 10 nucléotides en amont. Chez les eucaryotes, dans la très grande majorité des cas, la polymérase est positionnée par la fixation de la protéine TBP (en anglais, TATA-binding protein) à un court segment du promoteur contenant la séquence TATA (la « boîte TATA »), mais situé cette fois à 25 paires de bases en amont du point de départ. Au point de départ proprement dit se trouve un autre élément de signalisation, le site initiateur, mais dont la séquence est beaucoup moins conservée au cours de l'évolution. Une fois initiée, la transcription continue par élongation de l'ARN messager jusqu'au terminateur, situé juste après la région codante, et qui contient un signal de fin d'élongation.
5.2. La régulation de la transcription

À un moment donné, seuls certains gènes d’une cellule sont actifs et conduisent à la synthèse de protéines. Chez les organismes multicellulaires, les protéines produites dépendant du type de tissu ou d’organe. Par exemple, les cellules du derme de la peau synthétisent de la kératine, celles du pancréas, de l’insuline, celles des muscles squelettiques, des protéines contractiles, etc. Ainsi, les différents types cellulaires présentent chacun une combinaison particulière de gènes actifs, tandis que les autres restent inactifs. C’est la raison pour laquelle il doit nécessairement exister des molécules pour contrôler l’expression des gènes.
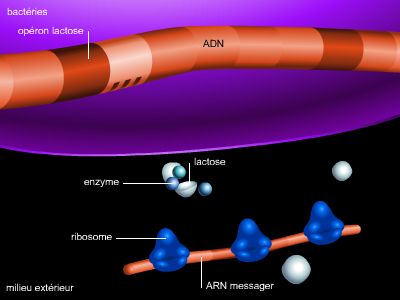
Cette tâche est dévolue aux régulateurs de transcription, que l'on peut classer en deux grands groupes : les répresseurs et les activateurs. Chacun contrôle toute une catégorie de gènes possédant dans la région régulatrice une même séquence de reconnaissance à laquelle il se lie spécifiquement. Chez les bactéries, la régulation est souvent fondée sur la répression : un répresseur est fixé sur son site de liaison, l'opérateur, situé à proximité du promoteur, gênant ainsi la fixation de la polymérase. La répression est levée par la présence d'un inducteur dans le milieu, qui agit en dissociant le répresseur de l'opérateur, permettant à la transcription de démarrer.
La situation est beaucoup plus complexe chez les eucaryotes, où le niveau de transcription d'un gène résulte de l'interaction globale de l'ensemble des activateurs et des répresseurs qui le contrôlent avec la machinerie de transcription. Les activateurs facilitent l'assemblage d'un complexe de préinitiation comprenant une dizaine d'entités multi-protéiques appelées « facteurs de transcription ». Les répresseurs empêchent l'ARN polymérase de démarrer la transcription. La transcription de chaque gène est contrôlée spécifiquement, chacun étant régulé par une combinaison unique d'activateurs et de répresseurs.
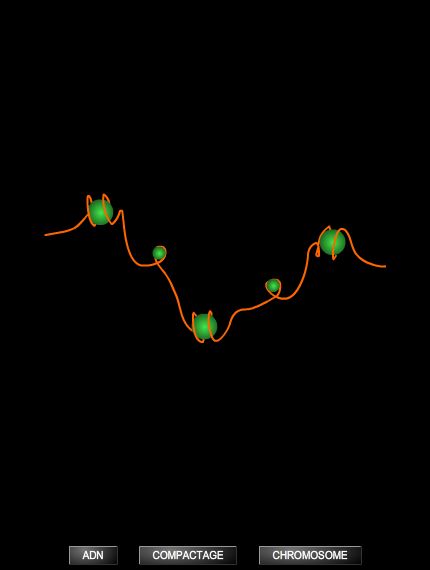
De plus, chez les eucaryotes, l'ADN est stocké de manière très compacte au sein du noyau sous forme de chromatine (forme désorganisée des chromosomes, qui apparaissent au moment de la mitose). Il s’agit d’une fibre en « collier de perles », dont les perles sont des nucléosomes. Ceux-ci sont constitués chacun d'un cœur de protéines, les histones, autour duquel la double hélice d'ADN est elle-même enroulée sur deux tours. Au sein de la chromatine, les facteurs de transcription et les protéines régulatrices n'ont pas accès aux gènes, ce qui est une façon de réprimer leur expression. Il faut l'intervention de protéines capables de remodeler la chromatine au niveau de ces sites régulateurs pour les rendre accessibles aux enzymes de transcription. L’observation de la chromatine montre de fait deux types d’organisation : l’euchromatine, très décondensée, correspond à des zones de gènes actifs, en cours de transcription ; l’hétérochromatine, plus condensée, renferme des gènes inactifs.